Obtenida la primera ‘película’ de la multiplicación del genoma del virus de la gripe
Investigadores de IMDEA Nanociencia han observado en directo, por primera vez, la multiplicación del genoma del virus de la gripe A. Este hecho ayudará a entender algunos de los factores que determinan la velocidad de multiplicación de este microorganismo infeccioso.
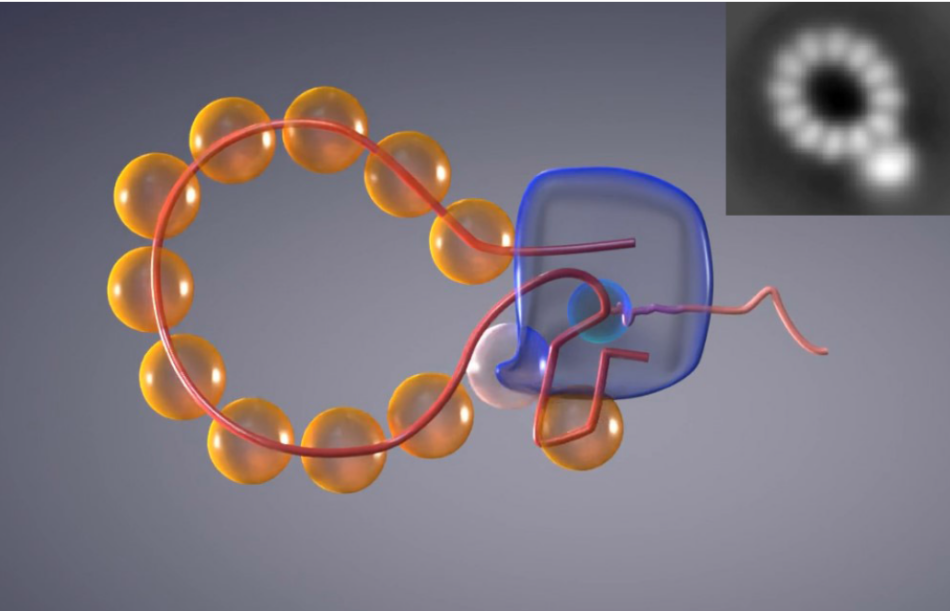
El virus de la gripe A es una gran amenaza que concierne a la salud pública. Comprender cómo se replica este virus es crucial, especialmente dado que sus mutaciones pueden dar lugar a nuevas cepas capaces de afectar a los humanos. En el núcleo del virus se encuentra la información genética, contenida en cadenas de ARN –ácido ribonucleico-, que la enzima polimerasa se encarga de copiar para generar nuevos virus.
Las cadenas de ARN están cubiertas por proteínas que protegen al ARN de ser degradado dentro de las células. ¿Cómo consigue la polimerasa multiplicar el ARN eficientemente si éste está totalmente cubierto de proteínas? Y además, ¿cómo consigue copiar el ARN sin desacoplarlo de las proteínas que lo protegen?
Durante el proceso de multiplicación del ARN, la polimerasa viral se desplaza a través de la estructura del ARN, sintetizando y copiando la estructura. Las proteínas que protegen el ARN del genoma del virus de la gripe A se organizan en forma de doble-hélice compacta, enmascarando la posición de la polimerasa.
Al no poder observar directamente la polimerasa en acción, muchos detalles del proceso de ‘copia’ se quedan ocultos sin poder ser observados. Hasta la fecha, no ha sido posible seguir el movimiento y la actividad de la polimerasa a largo del genoma del virus.
El grupo de investigación de ‘Manipulación de Motores Moleculares’ del Instituto Madrileño de Estudios Avanzados en Nanociencia, liderado por Borja Ibarra, en colaboración con investigadores delNanoLSI (Universidad Kanazawa Japón) y del Centro Nacional de Biotecnologia (CNB-CSIC) han ideado una estrategia que resulta clave para estudiar en detalle este elusivo proceso.
Los investigadores acortaron el genoma del virus para lograr que las proteínas que lo protegen se conformen en anillo, en lugar de una hélice. De esta forma, la posición de la polimerasa queda al descubierto.
Con esta estrategia, los investigadores pudieron analizar el movimiento de la polimerasa en tiempo-real, utilizando microscopia de fuerza atómica de alta velocidad. Grabaron múltiples películas del proceso “en directo”, que, combinando con imágenes de microscopía electrónica, les ayudaron a comprender y desvelar información novedosa sobre los procesos moleculares que gobiernan la amplificación del genoma viral.
Los investigadores observaron que la polimerasa se las arregla para acceder al ARN sin separarlo de las proteínas que lo protegen. Esto es esencial porque preserva la estructura del genoma, lo que a su vez, le permite multiplicarlo continuamente. La polimerasa es capaz de producir múltiples copias a partir del mismo ARN parental en varias rondas, lo cual es un aspecto clave para la multiplicación viral.
Un mecanismo de control en la multiplicación viral
Estas “películas” nanoscópicas permitieron a los investigadores estimar la tasa de síntesis de ARN, velocidad a la que trabaja la polimerasa viral. La polimerasa es capaz de incorporar hasta 35 nucleótidos en un segundo. Si equiparamos un nucleótido con una letra, un copista que trabajara a esta velocidad sería capaz de copiar la primera parte del Quijote en tan solo 6 horas (o el primer libro de Harry Potter en 3 horas).
El equipo de investigadores descubrió también que la estructura del ARN naciente condiciona la velocidad a la que trabaja la polimerasa. La conformación del ARN naciente funciona por tanto, como un mecanismo de control que regula la velocidad de amplificación del virus y podría suponer una diana terapéutica para el desarrollo de nuevas estrategias antivirales.
El ARN viral se multiplica rodeado de proteínas. A diferencia de trabajos anteriores que estudiaban la polimerasa aislada, este hallazgo se ha realizado en el entorno natural de la polimerasa, dentro del genoma y rodeada de proteínas con las que tiene que lidiar y que afectan a la velocidad de aplicación final.
El sistema modelo de este estudio proporciona una evidencia directa de que lasproteínas virales individuales pueden reciclarse, y confirma los modelos teóricos existentes. El trabajo ha sido bien recibido por la comunidad científica y ofrece un nuevo enfoque para investigar los mecanismos de transcripción y replicación viral en otros virus.
“Si conseguimos definir los mecanismos que gobiernan el funcionamiento de las proteínas virales, podremos idear métodos para interferir con ellos y, por tanto, parar la infección viral”, apuntaIbarra.
El trabajo, recientemente publicado en ACS Nano, sienta las bases para futuras investigaciones sobre funcionamiento de la polimerasa en el contexto del genoma viral, algo que hasta ahora no había sido posible.
Referencia:
Diego Carlero, Shingo Fukuda, Rebeca Bocanegra, Toshio Ando, Jaime Martin-Benito and Borja Ibarra. «Conformational dynamics of influenza A virus ribonucleoprotein complexes during RNA synthesis». ACS Nano.


